

一个R包本质上就是一系列函数的集合,通过调用R包的函数从而实现相应的功能。R包开发对R语言编程能力提升有极大帮助,更能方便理解他人编写的包,而不仅仅调参。
install.packages("usethis")
# install.packages("devtools")
devtools::install_github("r-lib/usethis")
一.创建一个R包¶
Rstudio中点击File - New Project - New Directory - R Package - 给自己R包取名和选择包地址,确定即可。


 通过代码创建
通过代码创建
devtools::create("F:/Rpkgs/YunSen")
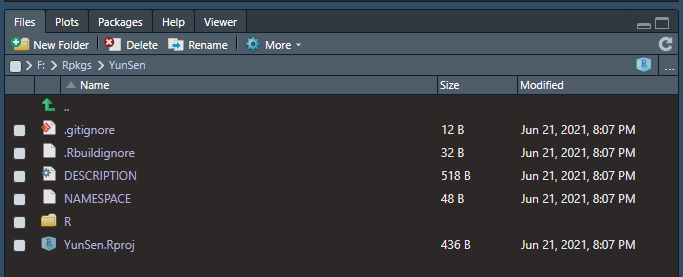 在包的文件下会生成5个文件和一个R文件夹
在包的文件下会生成5个文件和一个R文件夹
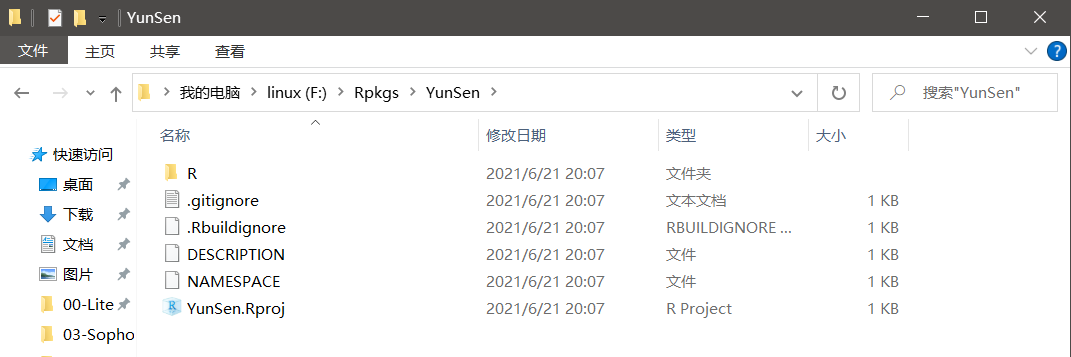 在RStudio中也可以看见。
在RStudio中也可以看见。
 R(存放R代码的地方)
DESCRIPTION(R包的相关描述)
NAMESPACE(用来规定可以调用的R函数功能,如果这里没有写明,即是R文件夹里面有相关函数功能也不能在之后调用)
.gitignore和.Rbuildgnore
R(存放R代码的地方)
DESCRIPTION(R包的相关描述)
NAMESPACE(用来规定可以调用的R函数功能,如果这里没有写明,即是R文件夹里面有相关函数功能也不能在之后调用)
.gitignore和.Rbuildgnore
二.创建R脚本¶
R脚本是存放在R这个文件夹下,但我们通常usethis工具创建。自动在R文件夹下生成脚本文件。
usethis::use_r("getSamInfo")
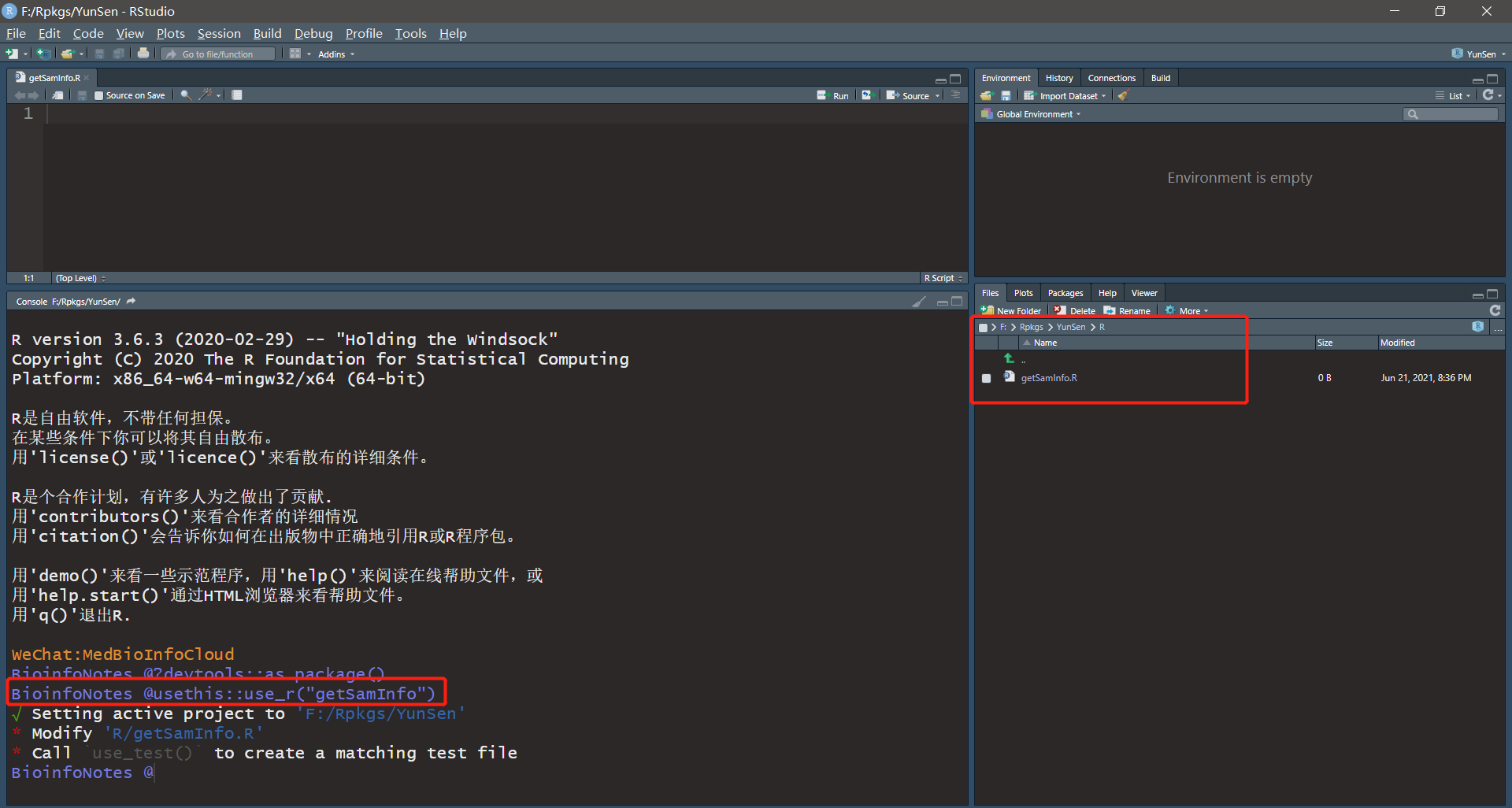 开始写脚本,代码一般全是函数。
开始写脚本,代码一般全是函数。
 写好脚本,我们要测试一下这个函数能否正常工作,首先使用load_all()函数加载包,它大致模拟了用library()安装和加载包时的情况。
写好脚本,我们要测试一下这个函数能否正常工作,首先使用load_all()函数加载包,它大致模拟了用library()安装和加载包时的情况。
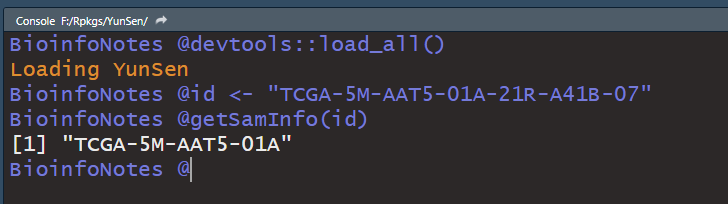 当我们把所有函数都写好后,利用devtools::check()自动检查构建和检查源代码包。
当我们把所有函数都写好后,利用devtools::check()自动检查构建和检查源代码包。
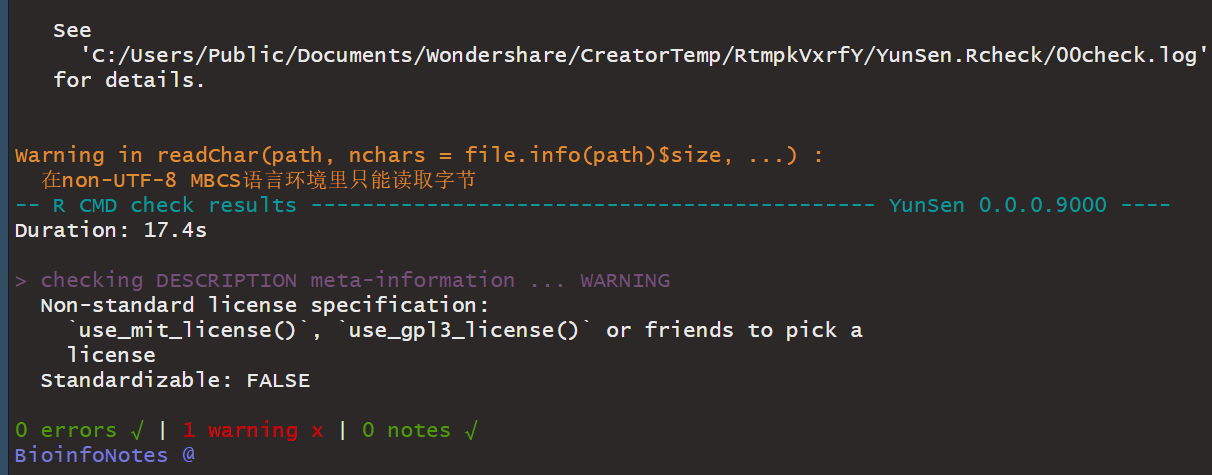 0个错误,1个警告,0个注意。可以看见警告内容是没有许可协议,如果不给其他人用,自己用的话,可以忽略许可协议。
怎么生成许可协议??首先就得介绍一下DESCRIPTION文件
0个错误,1个警告,0个注意。可以看见警告内容是没有许可协议,如果不给其他人用,自己用的话,可以忽略许可协议。
怎么生成许可协议??首先就得介绍一下DESCRIPTION文件
三.编写DESCRIPTION文件¶
DESCRIPTION文件的作用是存储包中重要的元数据,当创建包时,就默认生成了DESCRIPTION文件,默认已经有一些内容。
 Package:包的名字
Title: 包的介绍,这里的内容就是Rstudio中Packages面板下各种包的描述
Version: 0.0.0.9000(版本号,版本号存在一定规范。这里只是一个简单的R包开发教程,不需理会)
Author: 作者信息
Description: 包的描述
License: 许可证(包的公共许可,开源程序的特色)
Encoding: UTF-8(编码方式)
LazyData: true(R包内置数据时选中)
先修改作者信息。
Package:包的名字
Title: 包的介绍,这里的内容就是Rstudio中Packages面板下各种包的描述
Version: 0.0.0.9000(版本号,版本号存在一定规范。这里只是一个简单的R包开发教程,不需理会)
Author: 作者信息
Description: 包的描述
License: 许可证(包的公共许可,开源程序的特色)
Encoding: UTF-8(编码方式)
LazyData: true(R包内置数据时选中)
先修改作者信息。
 生成许可协议
生成许可协议
usethis::use_mit_license("Yun Sen")
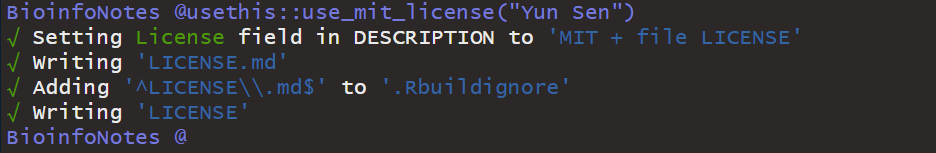 然后我们就会看见DESCRIPTION文件里的License处发生了变化
然后我们就会看见DESCRIPTION文件里的License处发生了变化
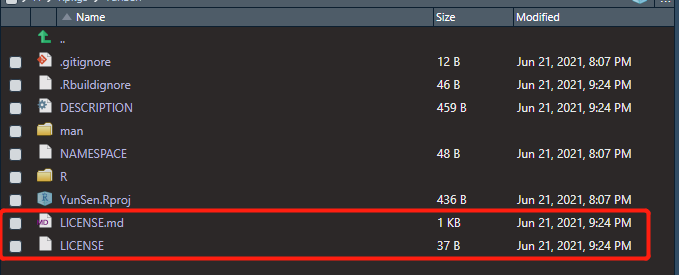
 同时在包目录下会生成2个文件。
同时在包目录下会生成2个文件。
四.写函数帮助文档¶
R 函数的帮助文档也是 R Package 的重要组成部分。函数文档以特殊的标记格式放在 man/.Rd 文件中,但是我们不需要掌握这种特殊标记语法,我们只需要撰写特殊格式的注释,然后 roxygen2 就会将其转换成 .Rd 文件。
将鼠标光标放在指定的函数体内,在 RStudio 中使用 Ctrl + Shift + Alt + R 或依次点击 Code -> Insert Roxygen Skeleton,就能够产生特殊格式的注释框架。
 然后会自动在该脚本前面生成默认的常见参数,都是以@开始的,这些我们可以称为标签
@param 函数参数
@return 说明本函数返回值
@importFrom 包名 函数名 函数名
@export(不添加,这个函数就不显示也就不能直接使用)
@examples提供示例代码
然后会自动在该脚本前面生成默认的常见参数,都是以@开始的,这些我们可以称为标签
@param 函数参数
@return 说明本函数返回值
@importFrom 包名 函数名 函数名
@export(不添加,这个函数就不显示也就不能直接使用)
@examples提供示例代码

 开始写参数介绍。
开始写参数介绍。
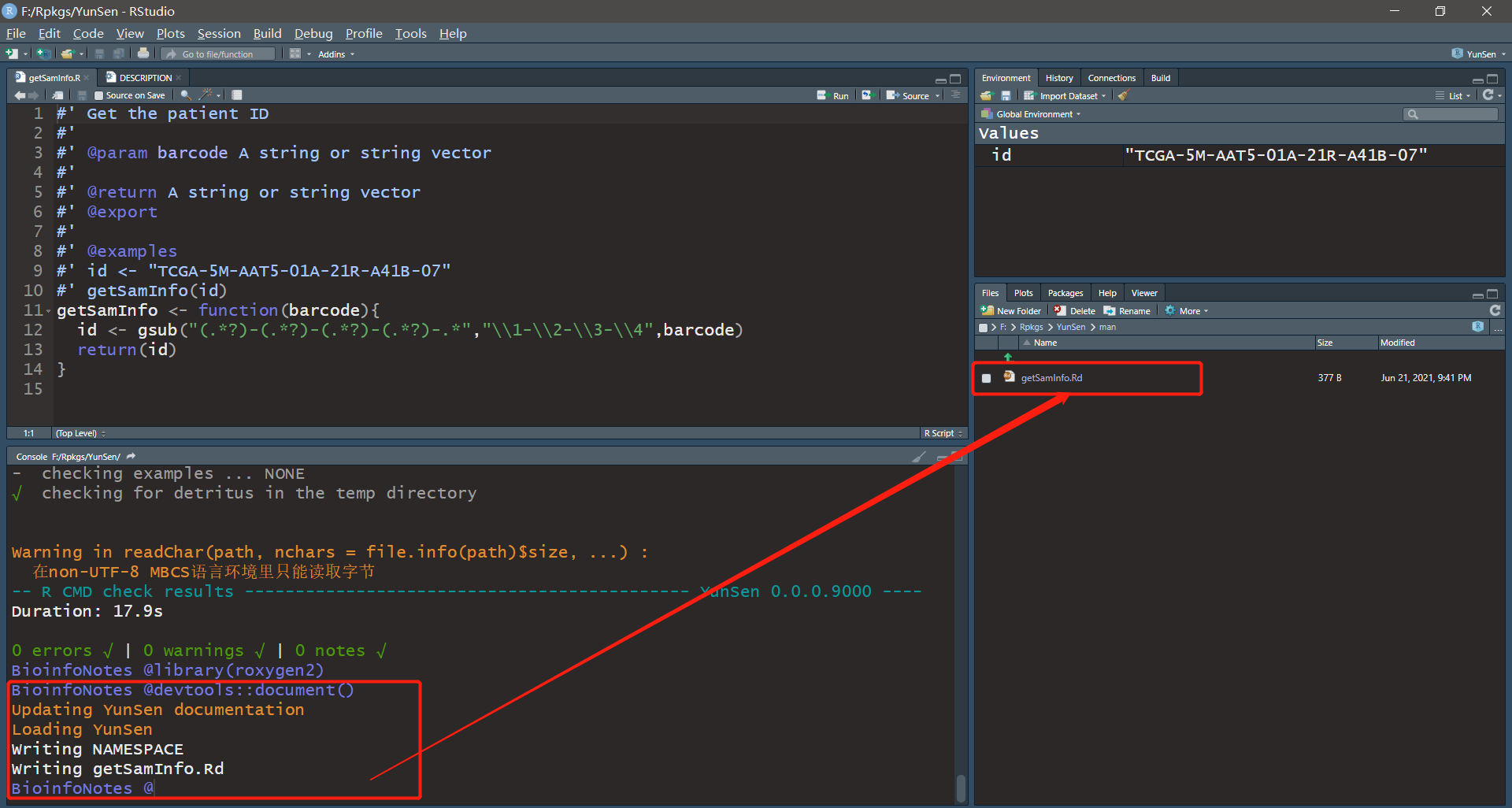
 document()函数生成帮助文档,执行devtools::document()后,会生成一个man文件夹,man文件夹下会生成相应的Rd文件,文件名就是函数名。
document()函数生成帮助文档,执行devtools::document()后,会生成一个man文件夹,man文件夹下会生成相应的Rd文件,文件名就是函数名。
devtools::document()
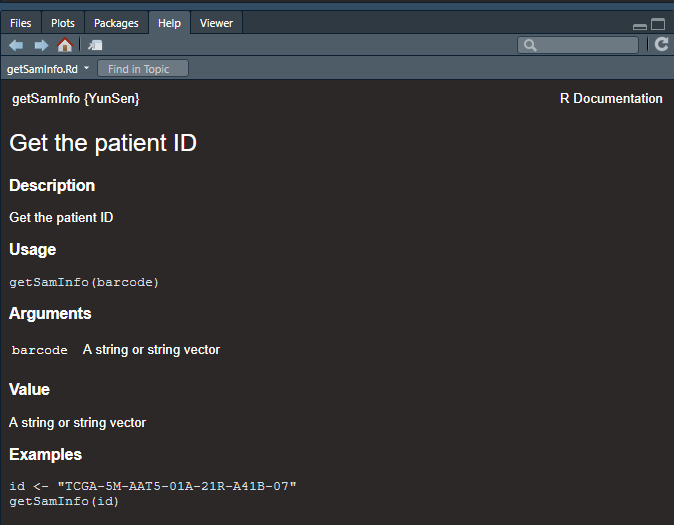
 然后我们就可以像平时一样通过问号?getSamInfo查看帮助文档了。
然后我们就可以像平时一样通过问号?getSamInfo查看帮助文档了。
五.添加测试代码¶
当我们写好一个函数后,需要验证函数是否能正确执行,结果是否正确,需要进行测试。
usethis::use_testthat()
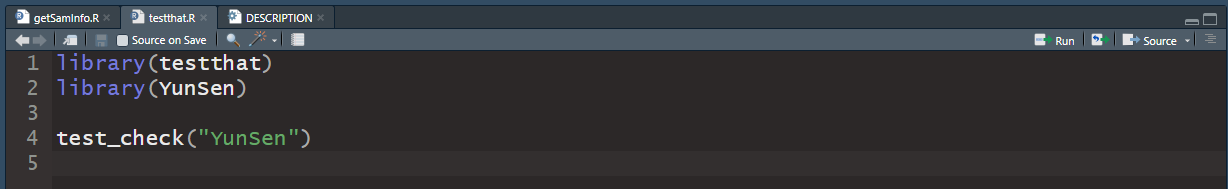
 test_check.文件里面的代码如下,就是加载我们自己的包和testthat包,和测试检查我们的包,这里先不管。
test_check.文件里面的代码如下,就是加载我们自己的包和testthat包,和测试检查我们的包,这里先不管。
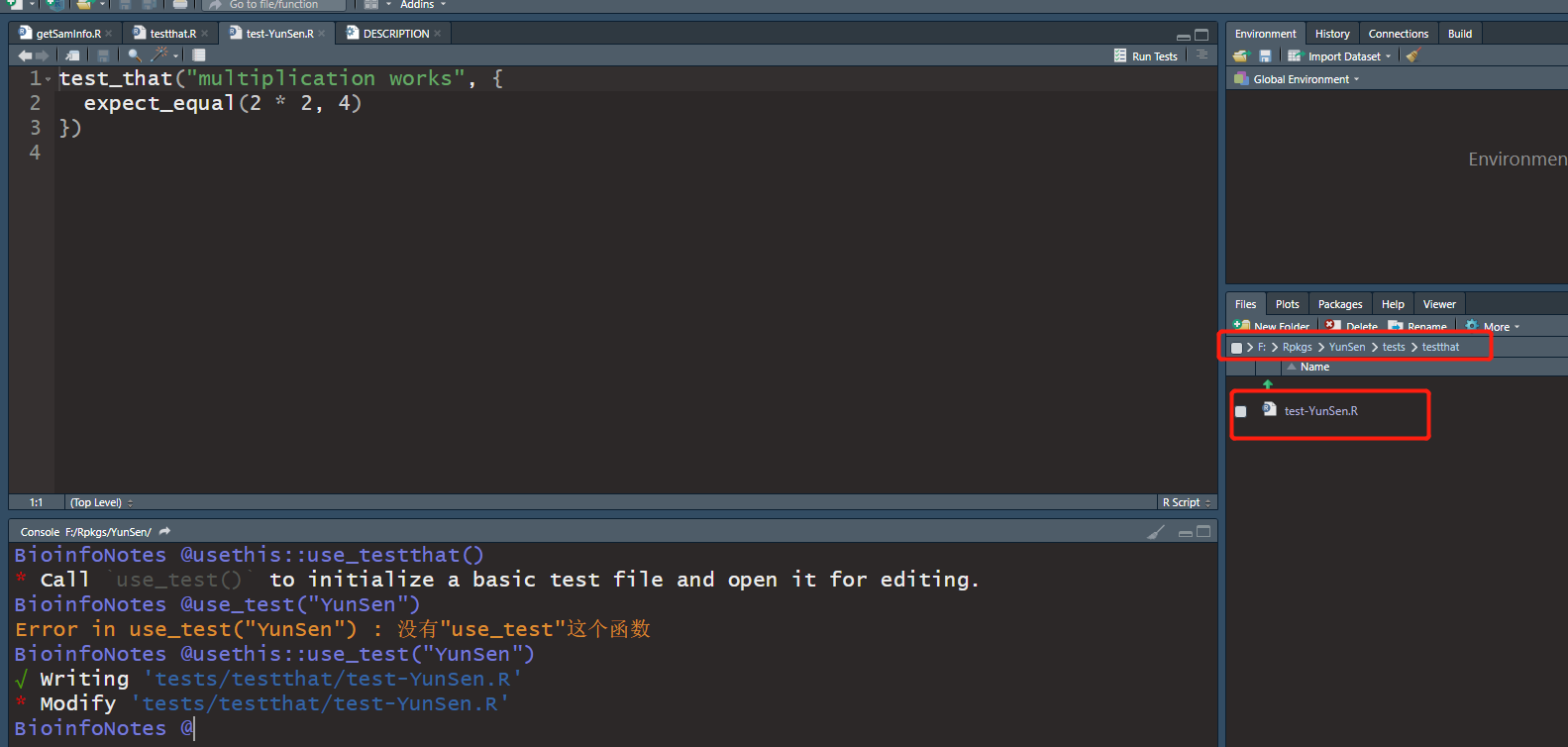
 我们要测试函数,需要自己创建一个R脚本,这里通过use_test函数创建,参数是包名
我们要测试函数,需要自己创建一个R脚本,这里通过use_test函数创建,参数是包名
usethis::use_test("YunSen")
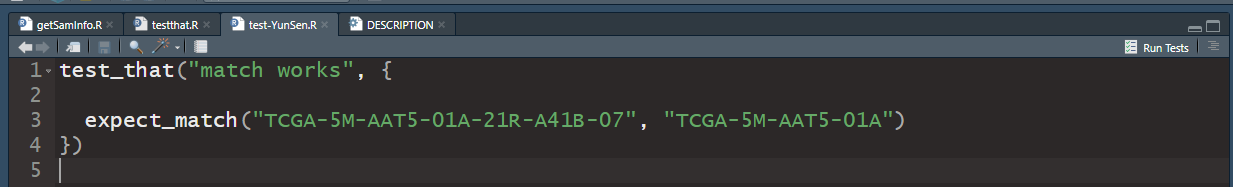
 我们主要是通过检查函数运行结果一预期结果的比较。通常会用到下面这些函数,但不是全部。
expect_equal()判断是否相等,它使用all.equal()来检查一个数值在误差内是否相等。expect_identical()精确测试是否相等。
expect_match检测一个字符串向量是否与一个正则表达式匹配。
expect_output()检测打印输出,expect_message()检查信息,expect_warning()检查警告,expect_error()检查错误。
我们主要是通过检查函数运行结果一预期结果的比较。通常会用到下面这些函数,但不是全部。
expect_equal()判断是否相等,它使用all.equal()来检查一个数值在误差内是否相等。expect_identical()精确测试是否相等。
expect_match检测一个字符串向量是否与一个正则表达式匹配。
expect_output()检测打印输出,expect_message()检查信息,expect_warning()检查警告,expect_error()检查错误。
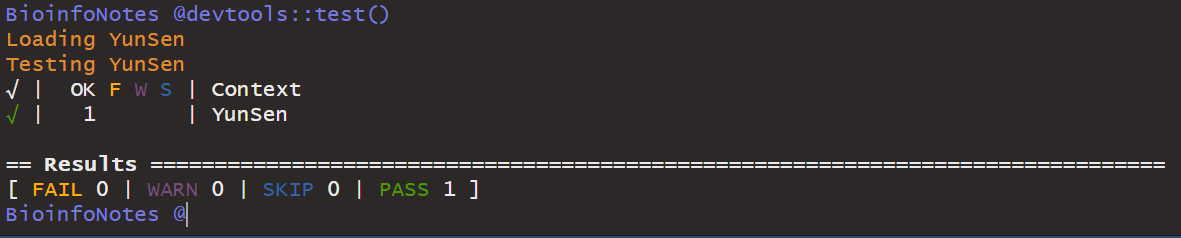
 然后执行devtools::test()进行检查
然后执行devtools::test()进行检查
devtools::test()
 如果这些函数不能满足我们的需要,我们需要用expect_true或者expect_false函数来抓取其他的期望。这里的期望是我自己去构造的,也就是构造一个返回值为true或者false的期望表达式。还有,有时候结果太复杂,不容易通过代码重建,在这种情况下,可以检查结果是否与上次相同,expect_equal_to_reference()会缓存第一次结果,然后将其与随后的运行进行比较。
如果这些函数不能满足我们的需要,我们需要用expect_true或者expect_false函数来抓取其他的期望。这里的期望是我自己去构造的,也就是构造一个返回值为true或者false的期望表达式。还有,有时候结果太复杂,不容易通过代码重建,在这种情况下,可以检查结果是否与上次相同,expect_equal_to_reference()会缓存第一次结果,然后将其与随后的运行进行比较。
六.依赖包¶
如果我们开发包过程中用到一些已有的包,或者其中的某个函数,那么我们需要引入,我们平时使用包是通过library或者source来加载包,但在开发时不能使用这种方式。R有很多种方式来描述这种潜在的依赖关系。通过imports(输入)、suggests(建议)、Depends(依赖)、LinkingTo(链接到)和Enhances(增强)来描述。imports里的包必须被安装,也就是在安装自己包的时候,imports申明的包也会被安装。suggests中的包,我们可以使用他,但不是必须的,比如我们可以退使用建议包中的数据来运行测试,编译使用指南,或者只有一个函数需要这个包,suggests中的包是不会随我们的包的安装而安装的。Depends用来指定特定的R版本,例如Depends:R(>=3.0.1)。LinkingTo用于列出依赖于另一个包中的C或者C++代码。Enhances列出你的包增强了的包,通常,你的包为其他包里的类提供了方法。 我们通过use_package()函数来申明依赖包。
use_package(package, type = "Imports", min_version = NULL)
usethis::use_package("GDCRNATools",type = "Imports")
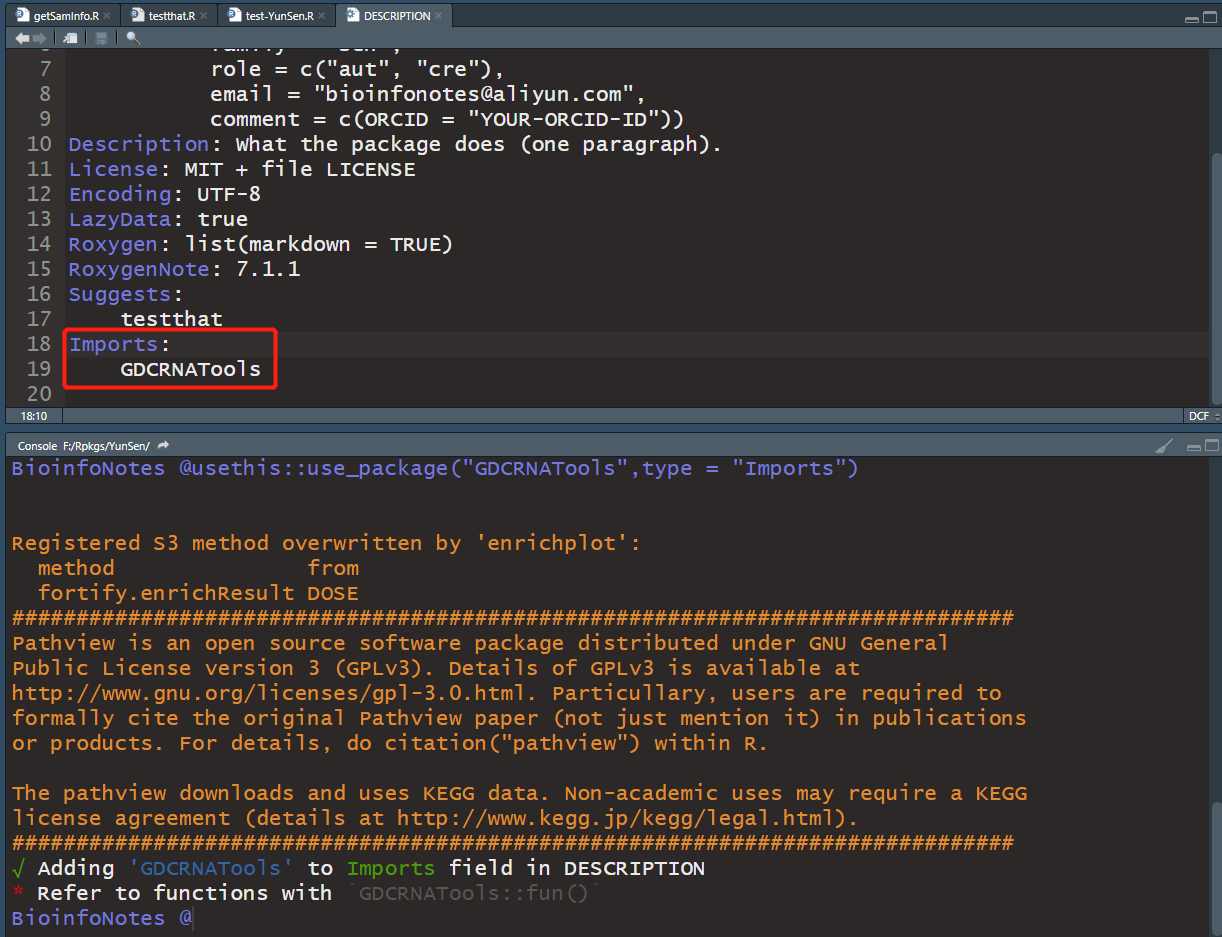 use_package()向DESCRIPTION添加了一个CRAN包依赖关系,并提供了一些关于如何最好地使用它的建议。use_dev_package()在一个正在开发的GitHub包上添加了一个版本化的依赖,将repo添加到Remotes,这样它就会从正确的位置自动安装。
我们这里创建一个新的R脚本
use_package()向DESCRIPTION添加了一个CRAN包依赖关系,并提供了一些关于如何最好地使用它的建议。use_dev_package()在一个正在开发的GitHub包上添加了一个版本化的依赖,将repo添加到Remotes,这样它就会从正确的位置自动安装。
我们这里创建一个新的R脚本
usethis::use_r("getTCGAData")
usethis::use_tidy_eval()
七. 外部数据¶
1.导出的数据¶
包中数据最常用的位置是在data/目录下,数据格式一般是.Rdata。其中包含单个对象和文件的名称相同,使用这个规则最简单的方法是使用:
usethis::use_data()
 会在包的目录下产生一个data文件夹,data目录下就会生成对应的Rda格式的文件。
会在包的目录下产生一个data文件夹,data目录下就会生成对应的Rda格式的文件。
 使用Use_data()可以很容易地以正确的格式保存包数据。我建议您将生成包数据的脚本保存在data-raw中:使用use_data_raw()进行设置。您还需要记录导出的数据集。
使用Use_data()可以很容易地以正确的格式保存包数据。我建议您将生成包数据的脚本保存在data-raw中:使用use_data_raw()进行设置。您还需要记录导出的数据集。
 在oneSamID写生成数据的代码,后续也好知道数据的来源,data-raw目录下的代码是不需要打包的。对于数据的描述文档可以使用下面2个标签描述。
在oneSamID写生成数据的代码,后续也好知道数据的来源,data-raw目录下的代码是不需要打包的。对于数据的描述文档可以使用下面2个标签描述。
' @format #' @source¶
2.内部数据¶
有时候函数需要预先计算的数据表,如果把这些数据放在data/目录下,这些数据也将会被提供给包的用户,这是不恰当的,正确的做法是把他保存在R、sysdata.Rda。我们在使用usethis::use_data()保存数据的时候,可以加上internal=TRUE来创建文件。

3.原始数据¶
如果想展示加载/解析原始数据的例子,就将原始文件放在inst/extdata。安装包时,inst/中所有文件和文件夹都会移动到顶层目录,所以它们不能有R/description这样的名字,要引用inst/extdata(无论是否安装)中是文件,使用system.file()。
 BioinfoNotes @use_data_table() √ Adding 'data.table' to Imports field in DESCRIPTION √ Writing 'R/utils-data-table.R' * Run
BioinfoNotes @use_data_table() √ Adding 'data.table' to Imports field in DESCRIPTION √ Writing 'R/utils-data-table.R' * Run devtools::document()
八.Build包¶
检查包
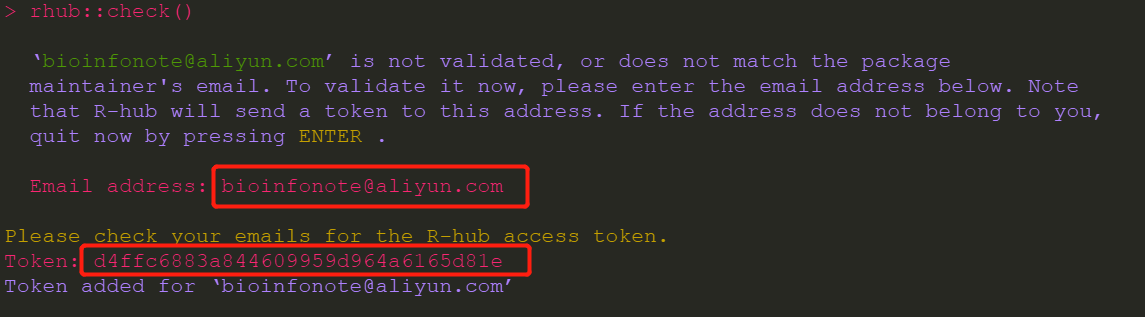
install.packages("rhub")
rhub::check()

九.提交到github¶
配置git用户: 在git窗口:
# configure the user which will be used by Git
# this should be not an acronym but your full name
git config --global user.name "Firstname Lastname"
# configure the email address
git config --global user.email "your.email@example.org"
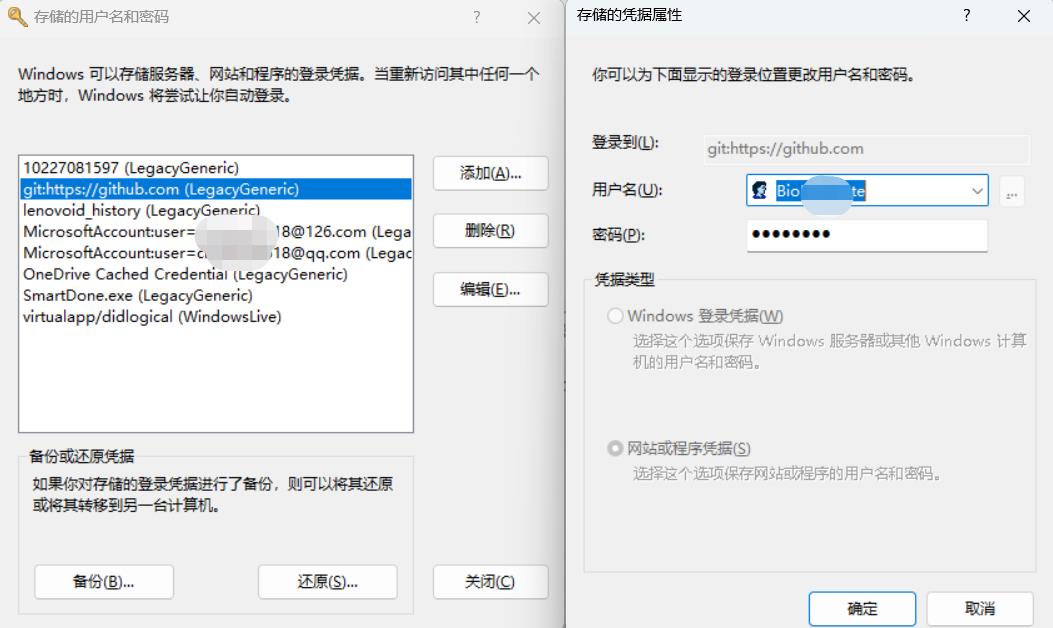 将 github 相关的条目删除。
然后提交
将 github 相关的条目删除。
然后提交
git remote add origin git@github.com:BioInfoNote/FUNscore.git
git push -u origin master
十. usethis包¶
1.use_package()¶
use_package()向DESCRIPTION添加了一个CRAN包依赖关系,并提供了一些关于如何最好地使用它的建议。 use_dev_package()添加了一个对正在开发的包的依赖,将dev repo添加到Remotes,这样它就会从正确的位置自动安装。 use_package(package, type = "Imports", min_version = NULL) use_dev_package(package, type = "Imports", remote = NULL) package:要依赖的包的名称。 type:依赖类型:必须是 "Imports", "Depends", "Suggests", "Enhances"h或者 "LinkingTo" 之一。匹配不区分大小写。 min_version:还可以为包提供一个最小版本。设置为TRUE以使用当前安装的版本。 remote:默认情况下,插入一个OWNER/REPO GitHub远程服务器。您可以选择提供一个字符串来指定远程,例如。"gitlab::jimhester/covr",使用远程包支持的任何语法。
2.use_import_from()¶
use_import_from(package, fun, load = is_interactive()) use_import_from()通过将roxygen2 @importFrom标记添加到包级文档(可以使用use_package_doc()创建)来从另一个包中导入函数。从另一个包中导入一个函数允许你在没有命名空间的情况下引用它(例如,fun()而不是package::fun())。use_import_from()也会重新记录命名空间,并重新加载当前包。这能够确保你的开发过程中能够立即获得乐趣。 package:Package name fun:A vector of function names load:Logical. Re-load with pkgload::load_all()?
3.use_r()¶
use_r(name = NULL, open = rlang::is_interactive()) use_test(name = NULL, open = rlang::is_interactive()) name:可以使用没有扩展名的名称,也可以使用NULL来根据脚本编辑器中当前打开的文件创建成对的文件。如果R文件是打开的,use_test()将创建/打开相应的测试文件;如果测试文件打开,use_r()将创建/打开相应的R文件。
4.use_rmarkdown_template()¶
use_rmarkdown_template( template_name ="Template Name", template_dir =NULL, template_description ="A description of the template", template_create_dir =FALSE)
5.Use_data()¶
use_data(..., internal =FALSE, overwrite =FALSE, compress ="bzip2", version =2)
library(GDCRNATools) samInfo <- gdcParseMetadata(project.id="TCGA-COAD", data.type='RNAseq') colnames(samInfo)
entity_submitter_id¶
TCGA-5M-AAT5-01A-21R-A41B-07¶
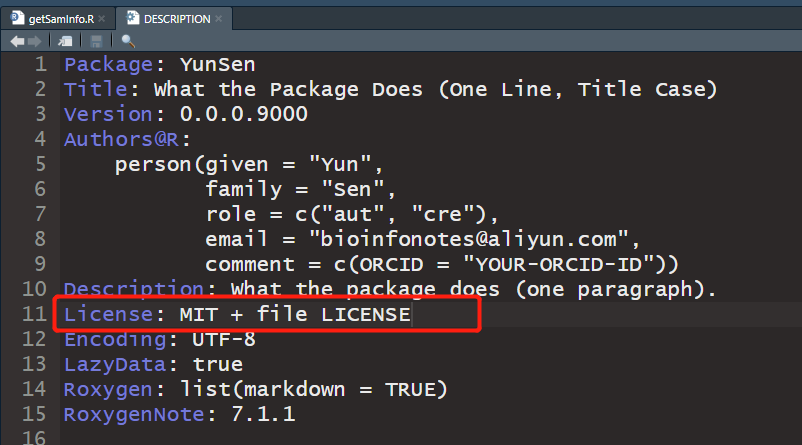
编写DESCRIPTION文件 在Rstudio中打开DESCRTION文件,编写包的描述。
相关内容包括: Package:包的名字 Type: Package(类型) Title: 包的介绍,这里的内容就是Rstudio中Packages面板下各种包的描述 Version: 0.1.0(版本号,版本号存在一定规范。这里只是一个简单的R包开发教程,不需理会) Author: 作者 Maintainer: The package maintainer yourself@somewhere.net(包的贡献者名字及邮箱) Description: 包的描述 License: 许可证(包的公共许可,开源程序的特色。这里也不理会,以GPL-3为协议) Encoding: UTF-8(编码方式) LazyData: true(R包内置数据时选中) Depends : 依赖环境 Imports : 需要使用的包 Suggests: 建议配合使用的包 其他如网址、bug报告地址等内容,这里不涉及。